Wie die IT in der Pharmakogenetik hilft, die Arzneimittelsicherheit zu erhöhen
Die Europäische Kommission schätzt, dass pro Jahr 197’000 PatientInnen an unerwünschten Arzneimittelwirkungen sterben. Dies liegt unter anderem auch an der unterschiedlichen genetischen Ausstattung – was bei Herrn Brönnimann gut wirkt, verfehlt bei Frau Brönnimann sein Ziel oder kann zu Nebenwirkungen führen. Dieser Einfluss lässt sich bereits vor der Medikamentengabe untersuchen, eine Anwendung des Instituts für Medizininformatik der Berner Fachhochschule unterstützt diesen wichtigen Prozess.
Die Pharmakogenetik (oft auch Pharmakogenomik/PGx genannt) untersucht den Zusammenhang zwischen den Genvariationen eines Patienten und seiner Verstoffwechselung von Arzneimitteln. Dieses Wissen erlaubt es, unerwünschte Arzneimittelwirkungen (UAW) bei Patienten mit bestimmten Genvariationen zu vermeiden. Aktuell sind vom Clinical Pharmacogenetics Implementation Consortium (CPIC) Guidelines zu 43 Wirkstoffen verfübar [1], darüberhinaus listet die FDA mittlerweile über 260 Wirkstoffe mit pharmakogenetischen Biomarkern im Beipackzettel [2].
Sowohl die Guidelines als auch die Biomarker werden bei der Verordnung und der Abgabe von Medikamenten derzeit noch selten beachtet. Dabei verursachen UAWs vermeidbares Leid und hohe Kosten im Gesundheitswesen. Die EU schreibt zu UAWs, respektive Adverse Drug Reactions (ADR):
It is estimated that 197,000 deaths per year in the EU are caused by ADRs and that the total cost to society of ADRs in the EU is €79 billion. Today’s proposals are aimed at further improving the current system. They will save many lives per year across the EU. In addition, they will help to cut red tape by decreasing the administrative burden by ca. € 145 billion p.a. – European Commission, 2008 [3]
Im Schweizerischen Gesundheitswesen fehlt es für die Nutzung der Potentiale von pharmakogenetischen Markern sowohl an den entsprechenden Anreizen als auch an effizienten und benutzerfreundlichen IT-Prozessen.
Wie wird Pharmakogenetik heute praktiziert?
Die Pharmakogenetik ist mittlerweile ein etabliertes Gebiet und die Pharmacogenomics Knowledge Base (PharmGKB) feiert schon bald ihren 20. Geburtstag. Pharmakogenetische Interaktionschecks sind schon seit einiger Zeit verfügbar und sind in den letzten Jahren immer günstiger geworden. Diese werden in der Schweiz von einigen Unternehmen angeboten und unterstützen in der Regel einen von zwei allgemein beschreibbaren Workflows.
Beim ersten Workflow wird eine entnommene Laborprobe der PatientInnen an das unternehmenseigene oder ein Partner-Labor gesandt, wo die Probe analysiert wird. Anschliessend wird aus den Labordaten ein Bericht erstellt, der entweder an die PatientInnen oder an die Behandelnden gesandt wird. Der übliche Transportweg ist dabei weiterhin die Post, wobei manche Anbieter auch den digitalen Versand via PDF-Datei anbieten. Die PatientInnen müssen selbst dafür sorgen, dass ihre Apotheke und behandelnden ÄrztInnen die pharmakogenetische Akte erhalten. Für Fachfremde schwer verständliche Berichte erfordern in manchen Fällen eine zusätzliche Konsultation mit FachärztInnen. Einige Unternehmen auf dem Markt haben sich darauf spezialisiert, aus den Labordaten möglichst verständliche Berichte zu erstellen, damit diese zusätzliche Konsultation eingespart werden kann.
Im zweiten Workflow gibt es ein Webportal, mit dem Laboraufträge erstellt und die dazugehörigen Analyseberichte abgerufen werden können. Statt nach der Laboranalyse und der darauf fussenden Berichterstellung den PatientInnen oder dem auftraggebenden Behandelnden nun einen Bericht zuzustellen, erhalten die PatientInnen einen digitalen Schlüssel, mit dem sie ihrer Apotheke oder ihren behandelnden ÄrztInnen den Zugriff auf die pharmakogenetischen Daten im Webportal gestatten können. Für die PatientInnen ist der Zugriff zum Bericht einfacher zu verwalten als in der ersten Variante.
Wieso ist die Pharmakogenetik trotz hoher Relevanz und existenter Anbieter bis heute noch wenig in der Praxis angekommen?
Ein Faktor für die mangelnde Berücksichtigung im Behandlungskontext ist die eher geringe Anzahl an Studien zum WZW-Nachweis (Wirksamkeit, Zweckmäßigkeit, Wirtschaftlichkeit) in der Anwendung von pharmakogenetischen Leitlinieninformationen. Daher verzichten noch viele Fachgesellschaften darauf, pharmakogenetische Interaktionschecks in den eigenen Leitlinien einzubinden. Man darf jedoch davon ausgehen, dass dies früher oder später geschehen wird.
Einige wenige pharmakologische Interaktionschecks dürfen bereits heute von ÄrztInnen aller Fachrichtungen in Auftrag gegeben und über die Krankenkasse abgerechnet werden. Dennoch kommen auch diese Tests bei vorhandener Indikation oft nicht zum Einsatz. Dies legt die Vermutung nahe, dass es noch weitere Hemmnisse gibt, beispielsweise der damit verbundene Aufwand. Wie oben angedeutet, existiert ein Mangel an integrativen IT-Services für die Verarbeitung und verständliche Darstellung von pharmakogenetischem Wissen. Dies hat zur Folge, dass eine effiziente Umsetzung und reibungslose Integration der pharmakogenetischen Prozesse in die bestehenden Prozesse oft schwierig zu realisieren ist.
Ein Lösungsansatz für die Zukunft
Das Institut für Medizininformatik der Berner Fachochschule hat eine Anwendung entwickelt, die sich über standardisierte Schnittstellen (Stichwort: HL7 FHIR) nahtlos in die Arbeitsprozesse der beteiligten Akteure einfügt und der Diversität von Prozessen Rechnung trägt. Die unterschiedlichen Dienste dieser Anwendung können entweder über ein Webportal verwendet oder in eine Drittsoftware integriert werden, beispielsweise in ein Praxisinformationssystem oder Apothekeninformationssystem. Die Anwendung kennt drei Prozessszenarien:
- Einen Initialprozess, wenn noch keine pharmakogenetischen Daten zum Patienten vorliegen.
- Die Durchführung eines pharmakogenetischen Interaktionchecks, wenn bereits pharmakogenetische Daten vorliegen.
- Das Abrufen eines pharmakogenetischen Berichts zum Patienten.
Der Initialprozess betrifft die erstmalige Durchführung eines pharmakogenetischen Interaktionschecks. Abbildung 1 gibt einen generischen Ablauf wieder, zu dem Variationen in der Anwendung existieren, um den unterschiedlichen Kontexten gerecht zu werden.
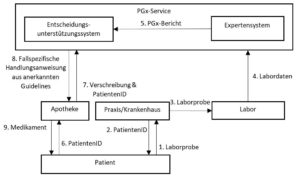
Abbildung 1: Workflow für eine pharmakogenetische Interaktionsprüfung mit initialer Genanalyse.
Dieser Prozess lässt sich an der Beispielpatientin Frau Brönnimann, die an Depressionen leidet und von Ihrem Psychiater Dr. Baumgartner Escitalopram verschrieben bekommen hat, veranschaulichen. Um sicherzugehen, dass Frau Brönnimann nicht länger als nötig auf die Rekonvaleszenz warten muss, schlägt Dr. Baumgartner Frau Brönnimann vor, einen pharmakogenetischen Interaktionscheck durchzuführen.
- Frau Brönnimann stimmt zu und Dr. Baumgartner entnimmt bei ihr eine Blutprobe für die Analyse (1).
- Der Laborauftrag wird über ein Webportal erfasst. Bei der Erfassung des Auftrages wird eine PatientenID generiert (2).
- Die Laborprobe wird ans Labor versandt (3),
- dort analysiert und die Labordaten an den PGx-Service übermittelt (4).
- Ein Expertensystem erstellt aus den Labordaten einen pharmakogenetischen Bericht (pharmakogenetisch relevante Genotypen von Frau Brönnimann) und übermittelt diesen ans Entscheidungsunterstützungssystem (5).
- Frau Brönnimann wird darüber informiert, dass die Daten analysiert wurden und ab sofort pharmakogenetische Interaktionschecks durchgeführt werden können. In der Apotheke gibt Frau Brönnimann der Apothekerin das Rezept und ihre PatientenID. Vor Abgabe des Medikaments werden diese Daten an das Entscheidungsunterstützungssystem gesendet (7).
- Dieses stellt mithilfe der hinterlegten Guidelines und dem PGx-Bericht fest, dass Frau Brönnimann ein «CYP2C19 Ultrarapid Metabolizer» ist. Dies bedeutet, dass Frau Brönnimann den Wirkstoff sehr viel schneller metabolisiert als andere Menschen, was wiederum dazu führt, dass die Konzentration des Wirkstoffes im Plasma sehr viel schneller unter das gewünschte Niveau fallen wird. Es besteht demnach eine hohe Wahrscheinlichkeit, dass Frau Brönnimann nicht auf das Medikament ansprechen wird [4,5].
- Diese Information übermittelt das Entscheidungsunterstützungssystem der Apothekensoftware zusammen mit einigen Vorschlägen für Alternativmedikamente, deren Metabolismus nicht primär mit dem Gen CYP2C19 zusammenhängt (8).
- Als die Apothekerin die Warnung auf dem Display sieht, greift sie zum Telefon und bespricht die Situation mit Dr. Baumgartner. Sie einigen sich darauf, dass Frau Brönnimann die Therapie mit einem der vorgeschlagenen Alternativmedikamente beginnt (9).
Dank des pharmakogenetischen Interaktions-checks konnte so verhindert werden, dass Frau Brönnimann ein Medikament einnimmt, das bei ihr mit hoher Wahrscheinlichkeit nicht wirkt und daher potenziell nur Nebenwirkungen erzeugt.
Drei Jahre später, die Depression ist schon halb vergessen, kommt Frau Brönnimann erneut in die Apotheke, um ihr neues Herz-Kreislauf Medikament zu bestellen. Da die benötigten Daten bereits vorhanden sind, kann das Entscheidungsunterstützungssystem vor Abgabe automatisch angestossen werden; es stellt kein erhöhtes Risiko fest. Es kann jederzeit auch ein klassischer pharmakogenetischer Bericht vom PGx-System bezogen werden.
Ausblick
Neben WZW-Studien zur Pharmakogenetik braucht es vermehrt Implementationsstudien, um zu zeigen, wie die Resultate aus der pharmakogenetischen Forschung der Bevölkerung effizient verfügbar gemacht werden können. Für Praxis-/Apotheken-/Krankenhaussoftwarehersteller muss es mit vertretbarem Aufwand möglich sein, pharmakogenetische Leitlinien und die notwendigen Zusatzprozesse in ihr System zu integrieren. Es sollte nicht an der IT scheitern, Patienten besser zu behandeln.
Literaturverzeichnis
- CPIC Guidelines [Internet]. [zitiert 11. Mai 2018].
- Research C for DE and. Table of Pharmacogenomic Biomarkers in Drug Labeling. FDA [Internet]. 27. März 2019 [zitiert 1. Mai 2019];
- European Commission – PRESS RELEASES – Press release – Strengthening pharmacovigilance to reduce adverse effects of medicines [Internet]. [zitiert 30. März 2019].
- Hicks J, Bishop J, Sangkuhl K, Müller D, Ji Y, Leckband S, u. a. Clinical Pharmacogenetics Implementation Consortium (CPIC) Guideline for CYP2D6 and CYP2C19 Genotypes and Dosing of Selective Serotonin Reuptake Inhibitors. Clinical Pharmacology & Therapeutics. August 2015;98(2):127–34.
- CPIC® Guideline for Selective Serotonin Reuptake Inhibitors and CYP2D6 and CYP2C19 [Internet]. [zitiert 25. Juli 2019].
 Create PDF
Create PDF

 Beiträge als RSS
Beiträge als RSS
Dein Kommentar
An Diskussion beteiligen?Hinterlasse uns Deinen Kommentar!